|
P-TEFb
![]() RNAمƒمƒھمƒ،مƒ©مƒ¼م‚¼IIمپ®ن¼¸é•·مپ®هˆ¶ه¾،م€پPol IIمپ¯è»¢ه†™é–‹ه§‹ç›´ه¾Œمپ«è² مپ®ن¼¸é•·ه› هگ(DSIFمپ¨NELF)مپ®هˆ¶ه¾،ن¸‹مپ«ç½®مپ‹م‚Œم‚‹م€‚P-TEFbمپ¯2مپ¤مپ®è² مپ®ه› هگمپ¨مƒمƒھمƒ،مƒ©مƒ¼م‚¼م‚’مƒھمƒ³é…¸م‚¢مپ™م‚‹مپ“مپ¨مپ§م€پç”ں産çڑ„مپھن¼¸é•·éپژ程مپ¸مپ®ç§»è،Œم‚’هھ’ن»‹مپ™م‚‹م€‚مپ“مپ®éپژ程مپ¯م€پ7SK snRNPمپ¨مپ®çµگهگˆمپ«م‚ˆمپ£مپ¦èھ؟節مپ•م‚Œم‚‹م€‚ RNAمƒمƒھمƒ،مƒ©مƒ¼م‚¼IIمپ®ن¼¸é•·مپ®هˆ¶ه¾،م€پPol IIمپ¯è»¢ه†™é–‹ه§‹ç›´ه¾Œمپ«è² مپ®ن¼¸é•·ه› هگ(DSIFمپ¨NELF)مپ®هˆ¶ه¾،ن¸‹مپ«ç½®مپ‹م‚Œم‚‹م€‚P-TEFbمپ¯2مپ¤مپ®è² مپ®ه› هگمپ¨مƒمƒھمƒ،مƒ©مƒ¼م‚¼م‚’مƒھمƒ³é…¸م‚¢مپ™م‚‹مپ“مپ¨مپ§م€پç”ں産çڑ„مپھن¼¸é•·éپژ程مپ¸مپ®ç§»è،Œم‚’هھ’ن»‹مپ™م‚‹م€‚مپ“مپ®éپژ程مپ¯م€پ7SK snRNPمپ¨مپ®çµگهگˆمپ«م‚ˆمپ£مپ¦èھ؟節مپ•م‚Œم‚‹م€‚
P-TEFb(positive transcription elongation factor b)مپ¯م€پçœںو ¸ç”ں物مپ«مپٹمپ„مپ¦RNAمƒمƒھمƒ،مƒ©مƒ¼م‚¼II(Pol II)مپ«م‚ˆم‚‹è»¢ه†™مپ®èھ؟節مپ«ه؟…è¦پن¸چهڈ¯و¬ مپھه½¹ه‰²م‚’وœمپںمپ™م‚؟مƒ³مƒ‘م‚¯è³ھ複هگˆن½“مپ§مپ‚م‚‹[1]م€‚مƒ’مƒˆéپ؛ن¼هگمپ®ه¤§éƒ¨هˆ†مپ§مپ¯م€پPol IIمپ¯è»¢ه†™مپ®é–‹ه§‹ç›´ه¾Œمپ«مƒ—مƒمƒ¢مƒ¼م‚؟مƒ¼è؟‘ه‚چمپ§مƒˆمƒ©مƒƒمƒ—مپ•م‚Œم‚‹[2][3]م€‚P-TEFbمپ¯م‚µم‚¤م‚¯مƒھمƒ³ن¾هکو€§م‚مƒٹمƒ¼م‚¼مپ§مپ‚م‚ٹم€پDSIF(英èھ版)(DRB sensitivity inducing factor)[4]مپ¨NELF(negative elongation factor)[5]م€پPol IIه¤§م‚µمƒ–مƒ¦مƒ‹مƒƒمƒˆمپ®Cوœ«ç«¯مƒ‰مƒ،م‚¤مƒ³ï¼ˆCTD)[6]م‚’مƒھمƒ³é…¸هŒ–مپ—م€پمپ“م‚Œمپ«م‚ˆمپ£مپ¦Pol IIمپ¯ç”ں産çڑ„(productive)مپھن¼¸é•·éپژ程مپ¸ç§»è،Œمپ—م€پmRNAمپ®هگˆوˆگمپŒè،Œم‚ڈم‚Œم‚‹م€‚P-TEFbمپ¯7SK snRNPمپ¨مپ®هڈ¯é€†çڑ„مپھçµگهگˆمپ«م‚ˆمپ£مپ¦éƒ¨هˆ†çڑ„مپ«èھ؟節مپ•م‚Œم‚‹[7]م€‚P-TEFbمپ®éک»ه®³ه‰¤مپ§مپ‚م‚‹DRB(英èھ版)م‚„مƒ•مƒ©مƒœمƒ”مƒھمƒ‰مƒ¼مƒ«ï¼ˆè‹±èھ版)مپ§ç´°èƒم‚’ه‡¦çگ†مپ™م‚‹مپ“مپ¨مپ§م€پmRNA産ç”ںمپ®ه–ھه¤±م€پوœ€çµ‚çڑ„مپ«مپ¯ç´°èƒو»مپŒه¼•مپچèµ·مپ“مپ•م‚Œم‚‹[6][8]م€‚
ç™؛見م€پو§‹وˆگمپ¨و§‹é€
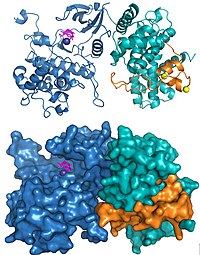 HIVمپ®TatمپŒçµگهگˆمپ—مپںP-TEFbمپ®و§‹é€ م€‚Cdk9مپŒé’م€پم‚µم‚¤م‚¯مƒھمƒ³T1مپŒم‚·م‚¢مƒ³م€پTatمپŒو©™م€پATPمپŒمƒم‚¼مƒ³م‚؟م€پن؛œé‰›هژںهگمپŒé»„色مپ§ç¤؛مپ•م‚Œمپ¦مپ„م‚‹م€‚PDB: 3MIA HIVمپ®TatمپŒçµگهگˆمپ—مپںP-TEFbمپ®و§‹é€ م€‚Cdk9مپŒé’م€پم‚µم‚¤م‚¯مƒھمƒ³T1مپŒم‚·م‚¢مƒ³م€پTatمپŒو©™م€پATPمپŒمƒم‚¼مƒ³م‚؟م€پن؛œé‰›هژںهگمپŒé»„色مپ§ç¤؛مپ•م‚Œمپ¦مپ„م‚‹م€‚PDB: 3MIA
P-TEFbمپ¯م€پم‚·مƒ§م‚¦م‚¸مƒ§م‚¦مƒگم‚¨ç´°èƒç”±و¥مپ®in vitro転ه†™ç³»م‚’用مپ„مپںé•·مپ„مƒ©مƒ³م‚ھمƒ•è»¢ه†™ï¼ˆè‹±èھ版)産物مپ®ه½¢وˆگمپ«ه؟…è¦پمپھه› هگمپ¨مپ—مپ¦هگŒه®ڑمپ•م‚Œم€پ精製مپ•م‚Œمپں[9]م€‚P-TEFbمپ¯م€پم‚·مƒ§م‚¦م‚¸مƒ§م‚¦مƒگم‚¨مپ§مپ¯è§¦هھ’م‚µمƒ–مƒ¦مƒ‹مƒƒمƒˆمپ§مپ‚م‚‹Cdk9مپ¨èھ؟節م‚µمƒ–مƒ¦مƒ‹مƒƒمƒˆمپ§مپ‚م‚‹م‚µم‚¤م‚¯مƒھمƒ³Tمپ‹م‚‰مپھم‚‹م‚µم‚¤م‚¯مƒھمƒ³ن¾هکو€§م‚مƒٹمƒ¼م‚¼مپ§مپ‚م‚‹[10]م€‚مƒ’مƒˆمپ®P-TEFbمپ«مپ¯è¤‡و•°مپ®ç¨®é،مپŒهکهœ¨مپ—م€پCdk9مپ¨مپ„مپڈمپ¤مپ‹مپ®م‚µم‚¤م‚¯مƒھمƒ³م‚µمƒ–مƒ¦مƒ‹مƒƒمƒˆم€پم‚µم‚¤م‚¯مƒھمƒ³T1م€پT2م€پKمپ®مپ†مپ،مپ®1مپ¤مپŒهگ«مپ¾م‚Œم‚‹[11][12]م€‚P-TEFbمپ¯مƒ–مƒمƒ¢مƒ‰مƒ،م‚¤مƒ³م‚؟مƒ³مƒ‘م‚¯è³ھBRD4م‚’هگ«م‚€ن»–مپ®ه› هگمپ¨çµگهگˆمپ—[13]م€پsuper elongation complexمپ¨ه‘¼مپ°م‚Œم‚‹ه·¨ه¤§مپھم‚؟مƒ³مƒ‘م‚¯è³ھ複هگˆن½“مپ«çµگهگˆمپ—مپ¦مپ„م‚‹مپ“مپ¨مپŒçں¥م‚‰م‚Œمپ¦مپ„م‚‹[14][15]م€‚é‡چè¦پمپھمپ“مپ¨مپ«م€پP-TEFbمپ¯HIVمپ®Tatم‚؟مƒ³مƒ‘م‚¯è³ھمپ®و¨™çڑ„مپ§مپ‚م‚ٹ[16]م€پTatمپ¯و£ه¸¸مپھç´°èƒه†…مپ®P-TEFbمپ®هˆ¶ه¾،م‚’è؟‚ه›مپ—مپ¦م€پHIVم‚²مƒژمƒ مپ®مƒ—مƒمƒ¢مƒ¼م‚؟مƒ¼è؟‘ه‚چمپ§هپœو¢مپ—مپںمƒمƒھمƒ،مƒ©مƒ¼م‚¼مپ«ç›´وژ¥P-TEFbم‚’م‚‚مپںم‚‰مپ™[17][18]م€‚
Cdk9مپ¨م‚µم‚¤م‚¯مƒھمƒ³T1م‚’هگ«م‚€مƒ’مƒˆP-TEFbمپ®و§‹é€ مپ¨HIV Tatمپ¨P-TEFbمپ®è¤‡هگˆن½“و§‹é€ مپŒXç·ڑçµگو™¶و§‹é€ 解وگمپ«م‚ˆمپ£مپ¦è§£مپ‹م‚Œمپ¦مپ„م‚‹م€‚وœ€هˆمپ«è§£مپ‹م‚Œمپںو§‹é€ مپ§مپ¯م€پ2مپ¤مپ®م‚µمƒ–مƒ¦مƒ‹مƒƒمƒˆمپŒن»–مپ®م‚µم‚¤م‚¯مƒھمƒ³ن¾هکو€§م‚مƒٹمƒ¼م‚¼مپ§è¦‹م‚‰م‚Œم‚‹م‚ˆمپ†مپھé…چç½®م‚’مپ—مپ¦مپ„م‚‹مپ“مپ¨مپŒç¤؛مپ•م‚Œمپں[19]م€‚ه…ƒم€…مپ®و§‹é€ مپ§مپ¯م‚µمƒ–مƒ¦مƒ‹مƒƒمƒˆمپ«3مپ¤مپ®و„ڈه›³مپ—مپھمپ„م‚¢مƒںمƒژ酸置وڈ›مپŒه°ژه…¥مپ•م‚Œمپ¦مپ„مپںمپŒم€پمپمپ®ه¾Œو£مپ—مپ„é…چهˆ—م‚’用مپ„مپ¦و§‹é€ و±؛ه®ڑمپŒè،Œم‚ڈم‚Œم€پو´»و€§éƒ¨ن½چه‘¨è¾؛مپ«مپ„مپڈمپ¤مپ‹ه¤§مپچمپھه¤‰هŒ–مپŒè¦‹م‚‰م‚Œمپںن»¥ه¤–مپ«مپ¯م€په…¨ن½“و§‹é€ مپ¯هگŒمپکمپ§مپ‚م‚‹مپ“مپ¨مپŒوکژم‚‰مپ‹مپ«مپ•م‚Œمپں[20]م€‚HIV TatمپŒP-TEFbمپ«çµگهگˆمپ—مپںو§‹é€ مپ‹م‚‰مپ¯م€پم‚¦م‚¤مƒ«م‚¹م‚؟مƒ³مƒ‘م‚¯è³ھمپ¯م‚µم‚¤م‚¯مƒھمƒ³T1م‚µمƒ–مƒ¦مƒ‹مƒƒمƒˆمپ¨ه؛ƒç¯„ه›²مپ«م‚ڈمپںم‚‹وژ¥è§¦é¢م‚’ه½¢وˆگمپ—مپ¦مپ„م‚‹مپ“مپ¨مپŒوکژم‚‰مپ‹مپ«مپ•م‚Œمپں[20]م€‚
P-TEFbمپ®èھ؟節
 P-TEFbمپ¨7SK snRNPمپ¨مپ®هڈ¯é€†çڑ„مپھçµگهگˆم€‚P-TEFbمپ¯Brd4مپ¾مپںمپ¯HIV Tatمپ«م‚ˆمپ£مپ¦7SK snRNPمپ‹م‚‰و”¾ه‡؛مپ•م‚Œم‚‹م€‚مپم‚Œمپ«ن¼´مپ£مپ¦HEXIMم‚‚除هژ»مپ•م‚ŒhnRNPمپŒ2مپ¤مپ®م‚؟مƒ³مƒ‘م‚¯è³ھمپ«ç½®مپچوڈ›م‚ڈم‚‹م€‚مپ“مپ®éپژ程م‚’هڈچ転مپ•مپ›م‚‹مپںم‚پمپ«مپ¯م€پن»–مپ®وœھçں¥مپ®ه› هگمپŒه؟…è¦پمپ§مپ‚م‚‹م€‚ P-TEFbمپ¨7SK snRNPمپ¨مپ®هڈ¯é€†çڑ„مپھçµگهگˆم€‚P-TEFbمپ¯Brd4مپ¾مپںمپ¯HIV Tatمپ«م‚ˆمپ£مپ¦7SK snRNPمپ‹م‚‰و”¾ه‡؛مپ•م‚Œم‚‹م€‚مپم‚Œمپ«ن¼´مپ£مپ¦HEXIMم‚‚除هژ»مپ•م‚ŒhnRNPمپŒ2مپ¤مپ®م‚؟مƒ³مƒ‘م‚¯è³ھمپ«ç½®مپچوڈ›م‚ڈم‚‹م€‚مپ“مپ®éپژ程م‚’هڈچ転مپ•مپ›م‚‹مپںم‚پمپ«مپ¯م€پن»–مپ®وœھçں¥مپ®ه› هگمپŒه؟…è¦پمپ§مپ‚م‚‹م€‚
P-TEFbمپ¯çœںو ¸ç”ں物مپ®éپ؛ن¼هگç™؛çڈ¾مپ®هˆ¶ه¾،مپ«ن¸ه؟ƒçڑ„مپھه½¹ه‰²م‚’وœمپںمپ™مپںم‚پم€پهگ„م‚µمƒ–مƒ¦مƒ‹مƒƒمƒˆم‚’م‚³مƒ¼مƒ‰مپ™م‚‹éپ؛ن¼هگمپ®è»¢ه†™م€پmRNAمپ®ç؟»è¨³م€پم‚؟مƒ³مƒ‘م‚¯è³ھمپ®م‚؟مƒ¼مƒ³م‚ھمƒ¼مƒگمƒ¼مپ®مƒ¬مƒ™مƒ«مپ§هژ³و ¼مپ«èھ؟節مپ•م‚Œمپ¦مپٹم‚ٹم€پمپ•م‚‰مپ«7SK snRNPمپŒé–¢ن¸ژمپ™م‚‹çڈچمپ—مپ„و©ںو§‹مپ«م‚ˆمپ£مپ¦م‚‚èھ؟節مپ•م‚Œمپ¦مپ„م‚‹[7][21]م€‚P-TEFbمپ¯ن؛Œوœ¬éژ–RNAçµگهگˆم‚؟مƒ³مƒ‘م‚¯è³ھHEXIM(مƒ’مƒˆمپ§مپ¯HEXIM1(英èھ版)مپ¾مپںمپ¯HEXIM2(英èھ版))مپ«م‚ˆمپ£مپ¦7SK snRNPمپ«ن؟وŒپمپ•م‚Œم‚‹م€‚7SK RNAمپ¾مپںمپ¯ن»–مپ®ن؛Œوœ¬éژ–RNAمپ«çµگهگˆمپ—مپںHEXIMمپ¯P-TEFbمپ«çµگهگˆمپ—م€پمپمپ®م‚مƒٹمƒ¼م‚¼و´»و€§م‚’éک»ه®³مپ™م‚‹[22][23]م€‚7SK RNAمپ«مپ¯ن»–مپ®2مپ¤مپ®م‚؟مƒ³مƒ‘م‚¯è³ھمپŒه¸¸مپ«çµگهگˆمپ—مپ¦مپ„م‚‹م€‚MEPCE(methylphosphate capping enzyme)مپ¯7SK RNAمپ®وœ€هˆمپ®مƒŒم‚¯مƒ¬م‚ھمƒپمƒ‰مپ®خ³-مƒھمƒ³é…¸مپ«مƒ،مƒپمƒ«هں؛م‚’ن»کهٹ مپ—[24]م€پLARP7(La-related protein 7)مپ¯7SK RNAمپ®3'وœ«ç«¯مپ«çµگهگˆمپ™م‚‹[25][26]م€‚7SK snRNPمپ‹م‚‰P-TEFbمپŒوٹœمپچهڈ–م‚‰م‚Œم‚‹مپ¨م€پ7SK RNAمپ¯م‚³مƒ³مƒ•م‚©مƒ،مƒ¼م‚·مƒ§مƒ³ه¤‰هŒ–م‚’èµ·مپ“مپ—م€پHEXIMمپ¯و”¾ه‡؛مپ•م‚Œمپ¦hnRNPمپŒé™¤هژ»مپ•م‚Œمپںه› هگمپ«ç½®مپچوڈ›م‚ڈم‚‹[7]م€‚P-TEFbمپŒه†چéڑ”離مپ•م‚Œم‚‹مپںم‚پمپ«مپ¯م€پRNAمپ®ه†چç·¨وˆگم€پHEXIMمپ®çµگهگˆم€پمپمپ—مپ¦مپمپ®ه¾Œمپ«P-TEFbمپ®çµگهگˆمپŒèµ·مپ“م‚‹ه؟…è¦پمپŒمپ‚م‚‹م€‚
ه‡؛ه…¸
- ^ Zhou, Qiang; Li, Tiandao; Price, David H. (2012). “RNA polymerase II elongation controlâ€. Annual Review of Biochemistry 81: 119–143. doi:10.1146/annurev-biochem-052610-095910. ISSN 1545-4509. PMC 4273853. PMID 22404626. https://pubmed.ncbi.nlm.nih.gov/22404626.
- ^ Rahl, Peter B.; Lin, Charles Y.; Seila, Amy C.; Flynn, Ryan A.; McCuine, Scott; Burge, Christopher B.; Sharp, Phillip A.; Young, Richard A. (2010-04-30). “c-Myc regulates transcriptional pause releaseâ€. Cell 141 (3): 432–445. doi:10.1016/j.cell.2010.03.030. ISSN 1097-4172. PMC 2864022. PMID 20434984. https://pubmed.ncbi.nlm.nih.gov/20434984.
- ^ Cheng, Bo; Li, Tiandao; Rahl, Peter B.; Adamson, Todd E.; Loudas, Nicholas B.; Guo, Jiannan; Varzavand, Katayoun; Cooper, Jeffrey J. et al. (2012-01-13). “Functional association of Gdown1 with RNA polymerase II poised on human genesâ€. Molecular Cell 45 (1): 38–50. doi:10.1016/j.molcel.2011.10.022. ISSN 1097-4164. PMC 3259526. PMID 22244331. https://pubmed.ncbi.nlm.nih.gov/22244331.
- ^ Wada, T.; Takagi, T.; Yamaguchi, Y.; Ferdous, A.; Imai, T.; Hirose, S.; Sugimoto, S.; Yano, K. et al. (1998-02-01). “DSIF, a novel transcription elongation factor that regulates RNA polymerase II processivity, is composed of human Spt4 and Spt5 homologsâ€. Genes & Development 12 (3): 343–356. doi:10.1101/gad.12.3.343. ISSN 0890-9369. PMC 316480. PMID 9450929. https://pubmed.ncbi.nlm.nih.gov/9450929.
- ^ Yamaguchi, Y.; Takagi, T.; Wada, T.; Yano, K.; Furuya, A.; Sugimoto, S.; Hasegawa, J.; Handa, H. (1999-04-02). “NELF, a multisubunit complex containing RD, cooperates with DSIF to repress RNA polymerase II elongationâ€. Cell 97 (1): 41–51. doi:10.1016/s0092-8674(00)80713-8. ISSN 0092-8674. PMID 10199401. https://pubmed.ncbi.nlm.nih.gov/10199401.
- ^ a b Marshall, N. F.; Peng, J.; Xie, Z.; Price, D. H. (1996-10-25). “Control of RNA polymerase II elongation potential by a novel carboxyl-terminal domain kinaseâ€. The Journal of Biological Chemistry 271 (43): 27176–27183. doi:10.1074/jbc.271.43.27176. ISSN 0021-9258. PMID 8900211. https://pubmed.ncbi.nlm.nih.gov/8900211.
- ^ a b c Peterlin, B. Matija; Brogie, John E.; Price, David H. (2012-01). “7SK snRNA: a noncoding RNA that plays a major role in regulating eukaryotic transcriptionâ€. Wiley interdisciplinary reviews. RNA 3 (1): 92–103. doi:10.1002/wrna.106. ISSN 1757-7012. PMC 3223291. PMID 21853533. https://pubmed.ncbi.nlm.nih.gov/21853533.
- ^ Chao, S. H.; Price, D. H. (2001-08-24). “Flavopiridol inactivates P-TEFb and blocks most RNA polymerase II transcription in vivoâ€. The Journal of Biological Chemistry 276 (34): 31793–31799. doi:10.1074/jbc.M102306200. ISSN 0021-9258. PMID 11431468. https://pubmed.ncbi.nlm.nih.gov/11431468.
- ^ Marshall, N. F.; Price, D. H. (1995-05-26). “Purification of P-TEFb, a transcription factor required for the transition into productive elongationâ€. The Journal of Biological Chemistry 270 (21): 12335–12338. doi:10.1074/jbc.270.21.12335. ISSN 0021-9258. PMID 7759473. https://pubmed.ncbi.nlm.nih.gov/7759473.
- ^ Peng, J.; Marshall, N. F.; Price, D. H. (1998-05-29). “Identification of a cyclin subunit required for the function of Drosophila P-TEFbâ€. The Journal of Biological Chemistry 273 (22): 13855–13860. doi:10.1074/jbc.273.22.13855. ISSN 0021-9258. PMID 9593731. https://pubmed.ncbi.nlm.nih.gov/9593731.
- ^ Fu, T. J.; Peng, J.; Lee, G.; Price, D. H.; Flores, O. (1999-12-03). “Cyclin K functions as a CDK9 regulatory subunit and participates in RNA polymerase II transcriptionâ€. The Journal of Biological Chemistry 274 (49): 34527–34530. doi:10.1074/jbc.274.49.34527. ISSN 0021-9258. PMID 10574912. https://pubmed.ncbi.nlm.nih.gov/10574912.
- ^ Peng, J.; Zhu, Y.; Milton, J. T.; Price, D. H. (1998-03-01). “Identification of multiple cyclin subunits of human P-TEFbâ€. Genes & Development 12 (5): 755–762. doi:10.1101/gad.12.5.755. ISSN 0890-9369. PMC 316581. PMID 9499409. https://pubmed.ncbi.nlm.nih.gov/9499409.
- ^ Yang, Zhiyuan; Yik, Jasper H. N.; Chen, Ruichuan; He, Nanhai; Jang, Moon Kyoo; Ozato, Keiko; Zhou, Qiang (2005-08-19). “Recruitment of P-TEFb for stimulation of transcriptional elongation by the bromodomain protein Brd4â€. Molecular Cell 19 (4): 535–545. doi:10.1016/j.molcel.2005.06.029. ISSN 1097-2765. PMID 16109377. https://pubmed.ncbi.nlm.nih.gov/16109377.
- ^ Smith, Edwin; Lin, Chengqi; Shilatifard, Ali (2011-04-01). “The super elongation complex (SEC) and MLL in development and diseaseâ€. Genes & Development 25 (7): 661–672. doi:10.1101/gad.2015411. ISSN 1549-5477. PMC 3070929. PMID 21460034. https://pubmed.ncbi.nlm.nih.gov/21460034.
- ^ He, Nanhai; Liu, Min; Hsu, Joanne; Xue, Yuhua; Chou, Seemay; Burlingame, Alma; Krogan, Nevan J.; Alber, Tom et al. (2010-05-14). “HIV-1 Tat and host AFF4 recruit two transcription elongation factors into a bifunctional complex for coordinated activation of HIV-1 transcriptionâ€. Molecular Cell 38 (3): 428–438. doi:10.1016/j.molcel.2010.04.013. ISSN 1097-4164. PMC 3085314. PMID 20471948. https://pubmed.ncbi.nlm.nih.gov/20471948.
- ^ Kao, S. Y.; Calman, A. F.; Luciw, P. A.; Peterlin, B. M. (1987 Dec 3-9). “Anti-termination of transcription within the long terminal repeat of HIV-1 by tat gene productâ€. Nature 330 (6147): 489–493. doi:10.1038/330489a0. ISSN 0028-0836. PMID 2825027. https://pubmed.ncbi.nlm.nih.gov/2825027.
- ^ Zhu, Y.; Pe'ery, T.; Peng, J.; Ramanathan, Y.; Marshall, N.; Marshall, T.; Amendt, B.; Mathews, M. B. et al. (1997-10-15). “Transcription elongation factor P-TEFb is required for HIV-1 tat transactivation in vitroâ€. Genes & Development 11 (20): 2622–2632. doi:10.1101/gad.11.20.2622. ISSN 0890-9369. PMC 316609. PMID 9334325. https://pubmed.ncbi.nlm.nih.gov/9334325.
- ^ Garber, M. E.; Wei, P.; Jones, K. A. (1998). “HIV-1 Tat interacts with cyclin T1 to direct the P-TEFb CTD kinase complex to TAR RNAâ€. Cold Spring Harbor Symposia on Quantitative Biology 63: 371–380. doi:10.1101/sqb.1998.63.371. ISSN 0091-7451. PMID 10384302. https://pubmed.ncbi.nlm.nih.gov/10384302.
- ^ Baumli, Sonja; Lolli, Graziano; Lowe, Edward D.; Troiani, Sonia; Rusconi, Luisa; Bullock, Alex N.; Debreczeni, Judit E.; Knapp, Stefan et al. (2008-07-09). “The structure of P-TEFb (CDK9/cyclin T1), its complex with flavopiridol and regulation by phosphorylationâ€. The EMBO journal 27 (13): 1907–1918. doi:10.1038/emboj.2008.121. ISSN 1460-2075. PMC 2486423. PMID 18566585. https://pubmed.ncbi.nlm.nih.gov/18566585.
- ^ a b Tahirov, Tahir H.; Babayeva, Nigar D.; Varzavand, Katayoun; Cooper, Jeffrey J.; Sedore, Stanley C.; Price, David H. (2010-06-10). “Crystal structure of HIV-1 Tat complexed with human P-TEFbâ€. Nature 465 (7299): 747–751. doi:10.1038/nature09131. ISSN 1476-4687. PMC 2885016. PMID 20535204. https://pubmed.ncbi.nlm.nih.gov/20535204.
- ^ Quaresma, AJ; Bugai A; Barboric M. (2016). “Cracking the control of RNA polymerase II elongation by 7SK snRNP and P-TEFb.â€. Nucleic Acids Research 44 (8): 7527–7539. doi:10.1093/nar/gkw585. PMC 5027500. PMID 27369380. http://nar.oxfordjournals.org/content/44/16/7527.long.
- ^ Li, Qintong; Cooper, Jeffrey J.; Altwerger, Gary H.; Feldkamp, Michael D.; Shea, Madeline A.; Price, David H. (2007). “HEXIM1 is a promiscuous double-stranded RNA-binding protein and interacts with RNAs in addition to 7SK in cultured cellsâ€. Nucleic Acids Research 35 (8): 2503–2512. doi:10.1093/nar/gkm150. ISSN 1362-4962. PMC 1885667. PMID 17395637. https://pubmed.ncbi.nlm.nih.gov/17395637.
- ^ Michels, Annemieke A.; Fraldi, Alessandro; Li, Qintong; Adamson, Todd E.; Bonnet, Franأ§ois; Nguyen, Van Trung; Sedore, Stanley C.; Price, Jason P. et al. (2004-07-07). “Binding of the 7SK snRNA turns the HEXIM1 protein into a P-TEFb (CDK9/cyclin T) inhibitorâ€. The EMBO journal 23 (13): 2608–2619. doi:10.1038/sj.emboj.7600275. ISSN 0261-4189. PMC 449783. PMID 15201869. https://pubmed.ncbi.nlm.nih.gov/15201869.
- ^ Jeronimo, Cأ©lia; Forget, Diane; Bouchard, Annie; Li, Qintong; Chua, Gordon; Poitras, Christian; Thأ©rien, Cynthia; Bergeron, Dominique et al. (2007-07-20). “Systematic analysis of the protein interaction network for the human transcription machinery reveals the identity of the 7SK capping enzymeâ€. Molecular Cell 27 (2): 262–274. doi:10.1016/j.molcel.2007.06.027. ISSN 1097-2765. PMC 4498903. PMID 17643375. https://pubmed.ncbi.nlm.nih.gov/17643375.
- ^ Krueger, Brian J.; Jeronimo, Cأ©lia; Roy, Bibhuti Bhusan; Bouchard, Annie; Barrandon, Charlotte; Byers, Sarah A.; Searcey, Courtney E.; Cooper, Jeffrey J. et al. (2008-04). “LARP7 is a stable component of the 7SK snRNP while P-TEFb, HEXIM1 and hnRNP A1 are reversibly associatedâ€. Nucleic Acids Research 36 (7): 2219–2229. doi:10.1093/nar/gkn061. ISSN 1362-4962. PMC 2367717. PMID 18281698. https://pubmed.ncbi.nlm.nih.gov/18281698.
- ^ He, Nanhai; Jahchan, Nadine S.; Hong, Eunmee; Li, Qiang; Bayfield, Mark A.; Maraia, Richard J.; Luo, Kunxin; Zhou, Qiang (2008-03-14). “A La-related protein modulates 7SK snRNP integrity to suppress P-TEFb-dependent transcriptional elongation and tumorigenesisâ€. Molecular Cell 29 (5): 588–599. doi:10.1016/j.molcel.2008.01.003. ISSN 1097-4164. PMC 6239424. PMID 18249148. https://pubmed.ncbi.nlm.nih.gov/18249148.
|
|